scRNAseq trouble shooting + etc (cellranger, scanpy, plotly)
토요일밤이다. 오늘은 논문읽는 속도가 좀 떨어진다. 항상 비슷한 패턴인데 평일날 열심히 공부하고, 실험하고 토요일은 반 정도 힘빼고 하는 것같다. 다음주 월요일에 저널미팅 발표가 있어서 내일은 PPT 준비해야 한다. 더 집중도 안되고해서 이번주 기록 남기고 자야겠다.
Trouble shooting + etc ..
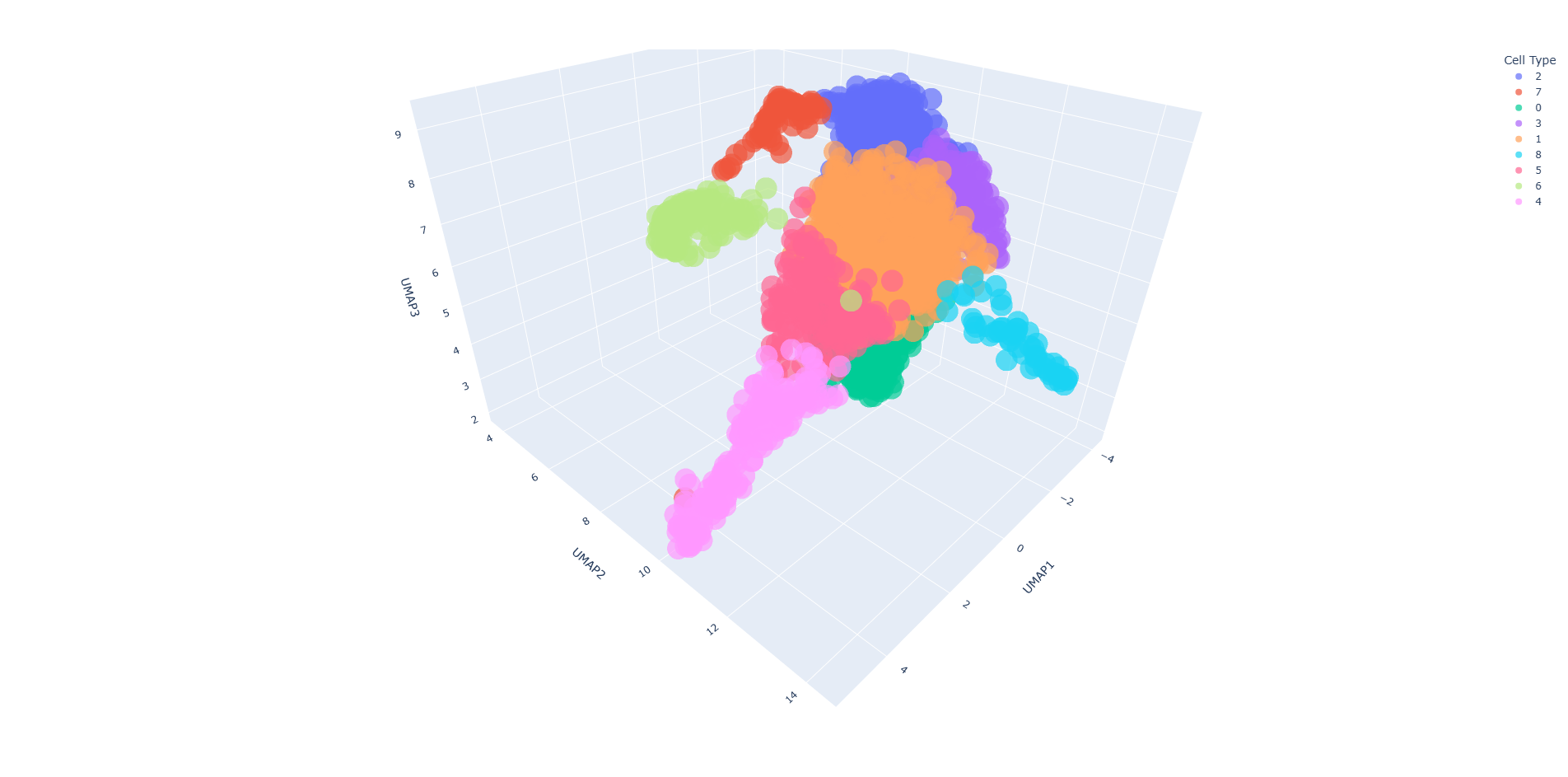
이건 약간 기믹인데 보통은 cell cluster를 UMAP1,2로 2차원으로 그리지만 UMAP3까지 3차원으로 그려봤다. 입체적으로 cell cluster 간의 거리를 더 직관적으로 파악할 수 있다. nature 논문에서 3차원 UMAP 을 보고 따라서 해본건데, 봤던 그림에선 cluster가 깔끔하게 나눠서 겹치는게 없었다. 내가 그린건 암 오가노이드처럼 생겼다.ㅋㅋ plotly로 그렸는데 옵션설정이 제한적이라 그림이 안이뻐서 그냥 2차원으로 돌아갔다.
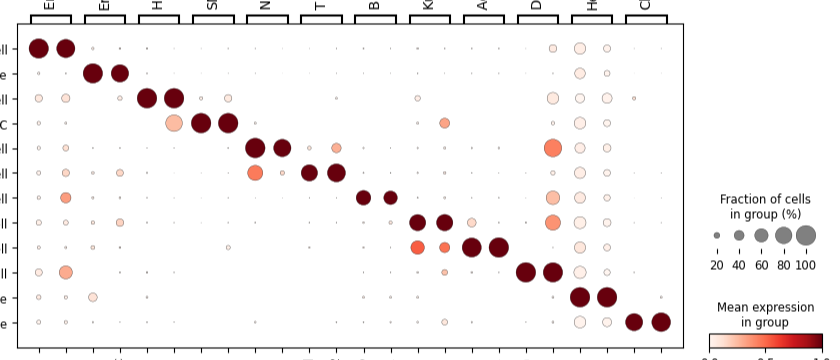
한달 반 정도 하니, 어느정도 논문에 나오는 figure 처럼 그림이 나온다. 실제 연구실 데이터로 만든거니 cluster와 gene 은 일단 다 잘랐다. 내부 데이터와 관련된 모든 것을 올리는데 조심스럽다.


같은 데이터 인데 cell ranger 결과가 극명하게 갈린다. cellranger version과 reference에 따라서 완전 다른데이터가 되버린다. 여기서 오는 딜레마가 있는데, 왼쪽의 최신 cellranger-9.0.0 와 최신 2024 transcriptomes reference를 이용한 결과가 QC 효율은 더 좋았지만(76.8%) 실제로 clustering 을 해보면 오른쪽(42.5%) 보다 잘 안되는 경향이 있다. 오른쪽 QC는 cellragner 6버전과 2020 reference를 이용하였다. 굉장히 당황스러운데, 어째서 더 좋은 QC 결과를 보인 최신판이 더 클러스터링이 안되는건지 이해를 할 수가 없다. 두개를 비교해본 결과 6버전 + 2020 에서는 total count (UMI)수 최저 threshold가 더 높게 잡혀 있었고 클러스터당 reads 수가 비교적 고르게 분포되어 있었다.다른 모양을 한 것처럼 보이지만 어차피 같은 데이터이고 threshold를 어떻게 주느냐에 따라서 clustering 결과가 달라지는 것이다.




차이를 볼 수 있는데 6버전에서 total counts 가 2100부근 이하가 댕강 잘려 나갔다. 9버전은 낮은 counts 부분을 포함하고 있는데 여기서 bias 꼈다고 추정하고 있다.
Genes detected in at least 3 cells were included. Cells that expressed fewer than 200 genes or had high mitochondrial genome transcript ratios (>0.2) were excluded. Single-Cell Transcriptomics Reveals Zone-Specific Alterations of Liver Sinusoidal Endothelial Cells in Cirrhosis
논문에서 찾은 QC조건인데 다음주에 gene, UMI threshold를 바꿔볼 예정이다. cellranger 돌리기전에 알고리즘 관련 옵션을 설정할 수 있는게 있는지 찾아보고 바꿔본다. low quality 를 판단하는 내부 알고리즘이 다를 것이라 추정하고 있다.
어이없는 실수를 했는데 파일을 잘못 넣었다..