| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | |||
| 5 | 6 | 7 | 8 | 9 | 10 | 11 |
| 12 | 13 | 14 | 15 | 16 | 17 | 18 |
| 19 | 20 | 21 | 22 | 23 | 24 | 25 |
| 26 | 27 | 28 | 29 | 30 | 31 |
- 대학생주식
- 통계학
- single cell rna sequencing
- 주식투자
- 선형대수
- Python
- Tutorial
- 후기
- R
- journal meeting
- scRNASeq
- numpy
- 대학원
- np.triu
- np.diagflat
- single cell rna sequening
- liver
- 연구원
- scanpy
- bioinformatics
- endothelial
- 주식
- 오블완
- 넘파이 매서드
- Preprocessing
- pcrnaseq
- 티스토리챌린지
- np.trace
- 대학생재테크
- np.flatten
- Today
- Total
biotechknowledge
생명정보개론(bioinformatics) - SNP, dbSNP 본문

이 포스트는 K-MOOC 의 생명정보개론 강의를 들으며 정리한 것으로 공부하면서 생겼던 의문점을 함께 정리하였습니다.
변이 데이터베이스
-2001년에 진행돼었던 human genome project 때 밝힌 인간의 DNA를 기준(reference)으로 삼는다.
SNP(Single nucleotide polymorphism)

DNA 염기서열에서 하나의 염기서열(A,T,G,C)의 차이를 보이는 유전적 변화 또는 변이를 단일 핵산염기 다형현상(SNP)라 한다. 여러사람들에게 공통적으로 나타난다면 (polymorphism) 이라 한다.

Q.어느 개인에게 SNP가 발견되었을 때, 그것이 질병을 일으키는 변이인지, 아니면 단순한 개인의 특징을 결정하는 변이인지 구분을 어떻게 하는가?
A. 데이터 베이스 참조 (ClinVar,dbSNP - 이미 알려진 질병 연관 변이를 포함하는 데이터베이스와 비교하여 구분한다.) 그 외 병리학적 예측 도구 이용, 유전자 기능과 변이 위치 분석 (SNP가 발생한 유전자의 역할과 증상 비교), 가족 연구(SNP가 가족 내에도 똑같이 발견 되는가? 똑같이 발견된다면 대립유전자 중 하나일 것), 실험적 검증(유전자 편집 기술을 이용한 세포실험, 동물실험), 발생 빈도, 임상 정보와 결합 비교 등이 있다.
dbSNP
이미 보고된 SNP를 모아 둔 데이터베이스. 실제 검색 예시 rs387907301 을 보자.


인종별로 정리된 SNP 빈도
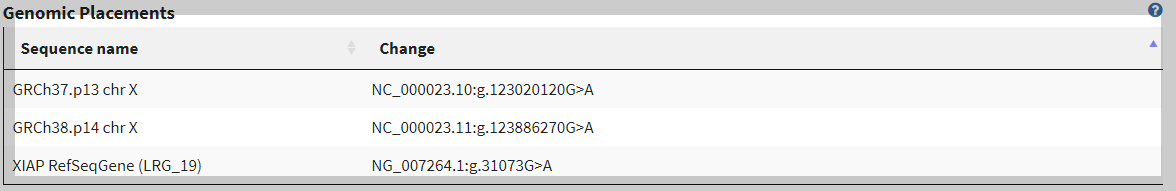
human genome도 계속 업데이트 되기 때문에 버전별로 SNP의 좌표가 앞뒤로 변동될 수 있다. 버전에 따른 SNP의 위치를 확인할 수 있다.
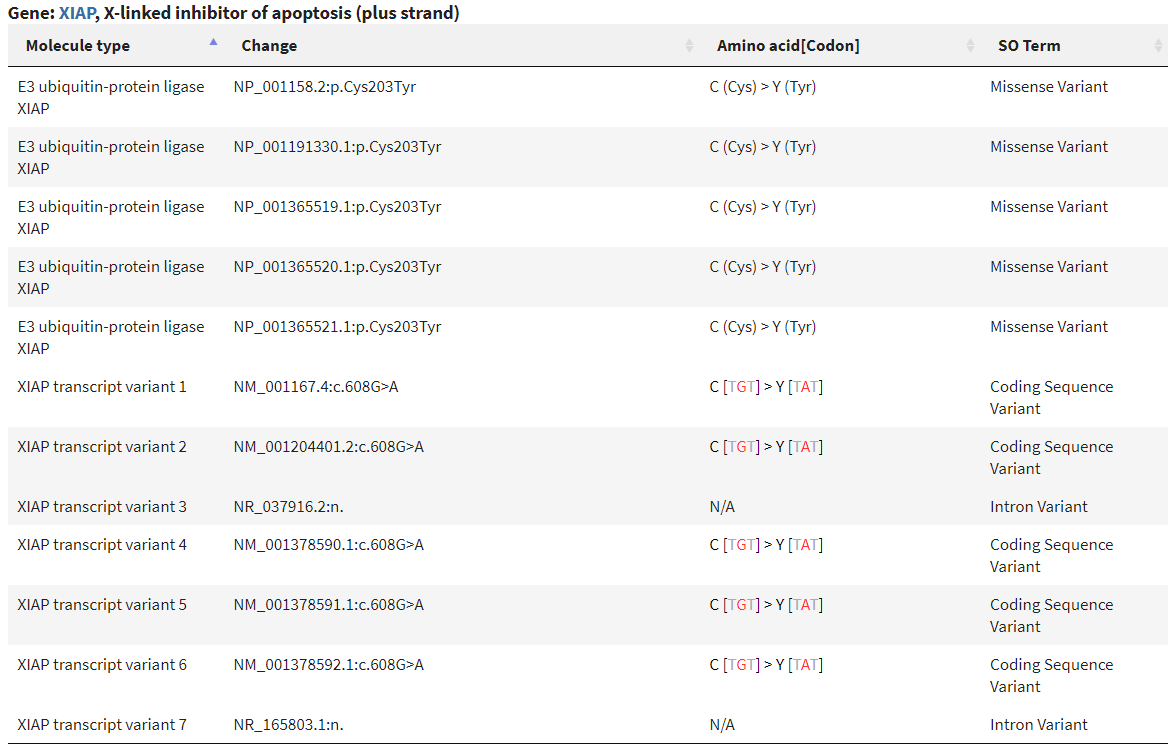
change 카테고리에 NP로 시작하는 것은 단백질에 생긴 돌연변이를 나타내고, NM으로 시작하는 것은 mRNA에 생긴 돌연변이를 나타낸다. NR은 인트론에 생긴 돌연변이를 나타낸다. XIAP 유전자는 5개의 mRNA SNP가 보고되었고 하나의 SNP당 하나의 아미노산을 변형 시켰다. (중복보함)
강의가 오래되어 UI와 표시된 정보가 달라서 이해하기 힘들다. 강의 내용으 충분하지 않아 다음에 따로 어떻게 읽는지 찾아보자.
'bioinformatics' 카테고리의 다른 글
| Scanpy tutorials - preprocessing and clustering ( Quality Control 개념설명) (4) | 2024.11.08 |
|---|---|
| Scanpy tutorials - preprocessing and clustering (anndata, pooch 개념설명) (2) | 2024.11.07 |
| 생명정보개론(bioinformatics) (0) | 2024.09.02 |
| 생명정보개론(bioinformatics) - Genebank,SNP 유전자 검색 (0) | 2024.08.14 |
| 생명정보개론(Bioinformatics)- NGS개념, 분석원리 (0) | 2024.08.13 |



