| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | ||
| 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| 13 | 14 | 15 | 16 | 17 | 18 | 19 |
| 20 | 21 | 22 | 23 | 24 | 25 | 26 |
| 27 | 28 | 29 | 30 |
- liver
- 선형대수
- scanpy
- Preprocessing
- np.flatten
- single cell rna sequening
- endothelial
- Python
- 후기
- 통계학
- numpy
- 넘파이 매서드
- 대학생재테크
- 대학생주식
- pcrnaseq
- journal meeting
- 대학원
- 티스토리챌린지
- 주식
- 오블완
- np.diagflat
- Tutorial
- np.trace
- bioinformatics
- single cell rna sequencing
- 주식투자
- singe cell ran sequencing
- np.triu
- R
- scRNASeq
- Today
- Total
biotechknowledge
[Journal review]Paired-cell sequencing enables spatial gene expression mapping of liver endothelial cells 저널 리뷰/ 발표자료 본문
[Journal review]Paired-cell sequencing enables spatial gene expression mapping of liver endothelial cells 저널 리뷰/ 발표자료
준2준2 2024. 12. 16. 16:50
nature biotechnology에 실린 pair-cell sequencing을 이용한 liver endothelial heterogeneity 를 연구한 내용이다. single-cell RNA seq은 cell subtype 연구에 필수불가결한 아주 강력한 데이터이다. 하지만 cell 의 위치정보는 잃어버린다는 단점이 있다. 이 단점을 극복하기 위한 방법으로서 paried-cell 을 sequencing하는 방법을 제시하고 있고 biology 적인 내용보다 실험테크닉에 의미를 두고 있고 그래서 biotechnology에 실린 것 같다.


일반적인 scRNAseq flow이다. barcode 1개에 cell 1개를 붙여 droplet을 만들고 (한 방울) sequencing 한다. 이 논문에서는 이 하나의 bead 에 두개의 cell을 같이 sequencing을 한다. 이해를 돕기위해 오른쪽 그림을 보면 빨강색(EC, endothelial cell), 파란색(hepatocyte, 간세포) 를 그림으로 표시해봤다. EC+hepatocyte 가 1개의 sample, 즉 table의 한 행이되는 것이다.

paried-cell을 이용한 배경에 대해 이해할 필요가 있다. 간세포(hepatocyte)는 heterogeneity가 있다고 학계에 일관적인 consensus가 있었고 EC는 세포수가 적고 sparsity가 심해서 연구가 잘 안되고 있었다. 이 연구팀은 앞서 naure 에 출판한 hepatocyte gene atlas 를 가지고 있고 이 데이터와 pcRNAseq 데이터를 이용해서 EC 특이적인 gene panel을 찾아낸 내용을 담고 있다.


그림과 함께 다시 설명하자면, 벤다이어그램이 하나의 sample에 속해있는 mRNA library이다. sample에는 hepatocyte 유전자와 EC 유전자가 섞여있다. 연구팀은 Hepatoctye 의 gene atlas를 완벽하게 구축하는데 성공하였고 nature에 출판하였다. 이 데이터를 전체에서 차집합하면 endothelial specific gene을 찾아내는 과정이다.
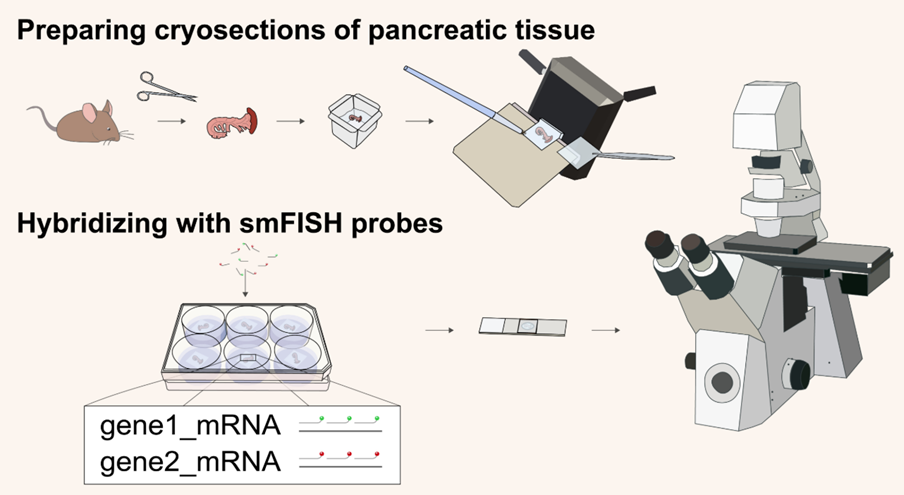
논문에서 insilco data를 검증하기 위해 smFISH 실험기법을 이용한다. confocal 과 비슷한데 sample prep 해서 section 하고 항체대신 mRNA probe 형광 prob를 붙인다. confocal 에서는 형광항체를 붙여 단백질 수준의 phenotype 을 확인하는 것이라면 smFISH에서는 mRNA level 의 시각화한다는 것에 차이점이 있다.
논문의 내용을 크게 세 파트로 나눠보자면 1.paired-cell selection and validation, technology의 핵심인 paired-cell을 걸러낸 방법과 그 검증에 대해서 다루고 2. scRNAseq 데이터에서 hepatoctye 유전자를 제거하고 EC 특이적인 유전자를 추출해내는 과정과 3. 추출해낸 EC genes 을 정리하고 발견사항을 정리하는 과정으로 나눌 수 있다.
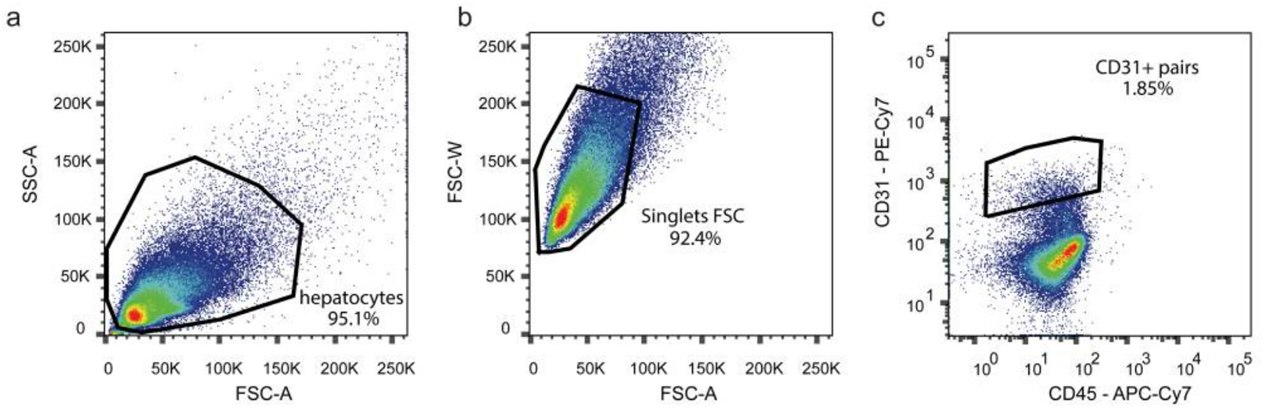
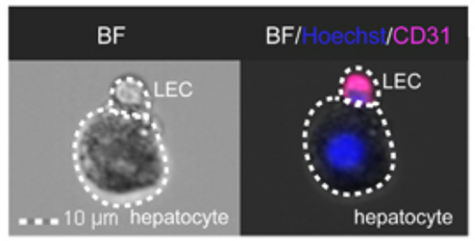
doublet cell(hepatocyte +EC) 를 모으기 위해 hepatocyte gating을 한 후 CD31(EC marker)를 이용해서 gating 한다. 여기서 b에서 singlets 을 gating 했는데 doublet cell(hepatocyte +EC)가 걸러지지 않을 수 있던 이유는 오른쪽 visual image를 보면 hepatoctye가 LEC 보다 몇배 더 큰걸 확인할 수 있다. EC가 충분히 작기 때문에 singlet gating에 통과한 것으로 추정할 수 있다.
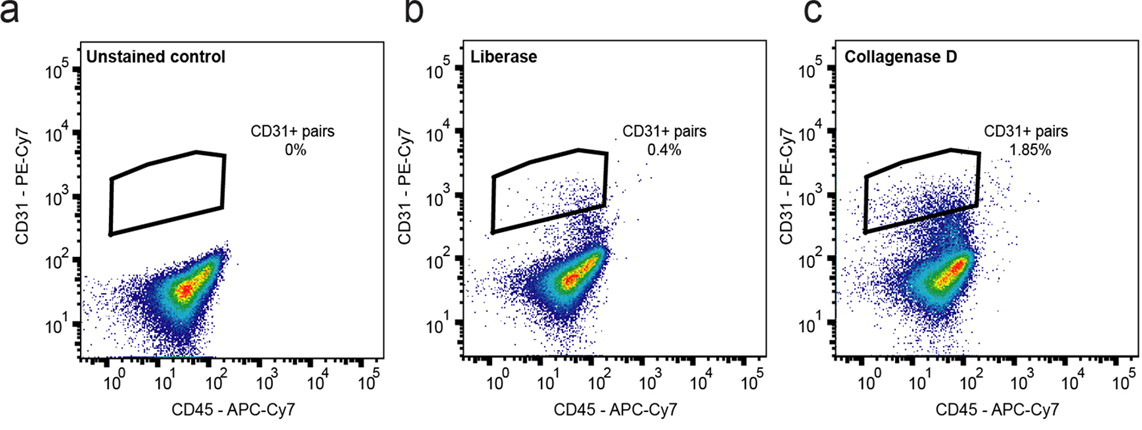
연구팀의 노하우인데, sample prep agent를 바꿔가면서 일부러 cell dissociation 효율이 떨어지는 것을 찾는 것이다. 왜냐면 실험의 목적상 hepatocyte 와 EC 가 붙어있는것들을 모아야 하기 때문이다. 연구팀에서 Collagenase D를 사용해였다.
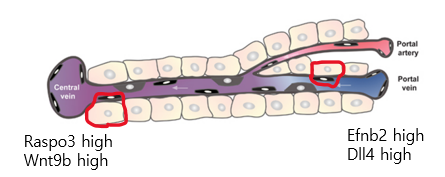
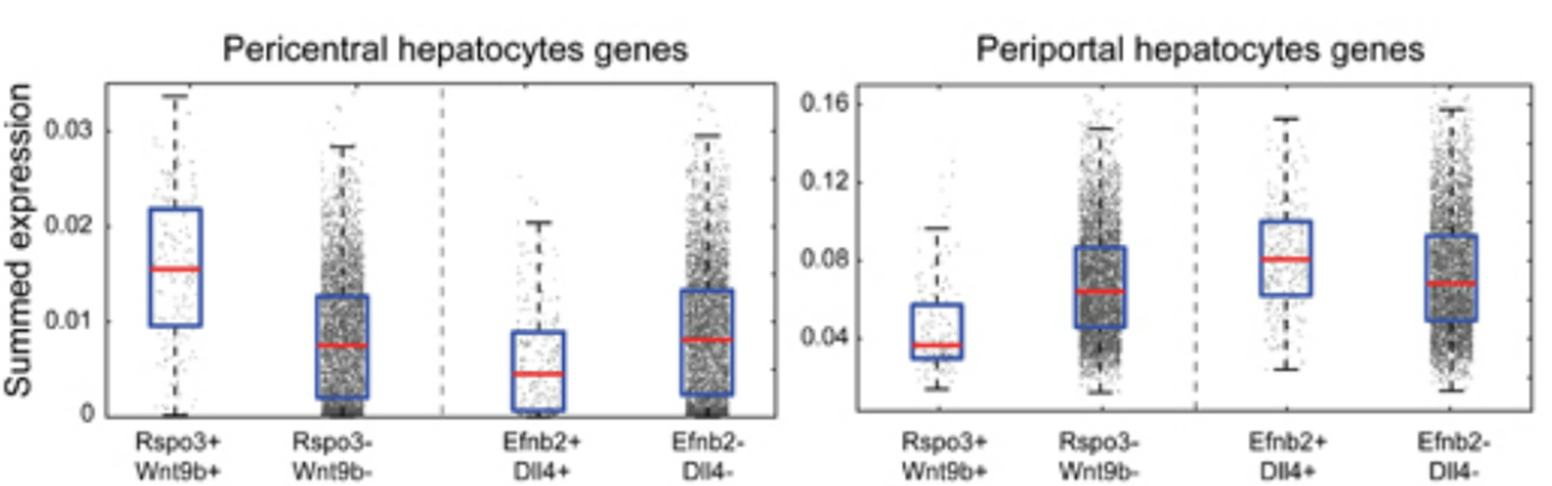
왼쪽 그림은 pcRNAseq 데이터를 이루는 하나의 sample을 보여주는 것이다. 오른쪽에서 Rspo3, Wnt9b는 endothelial cell Central vein 에서 높은 발현을 보이는게 알려져 있는 유전자이고 Efnb2, Dll4는 Portal vein에서 높을 발현을 보이는게 알려져 있는 유전자이다. hep cell 과 EC 가 섞여있는 pcRNAseq sample 갖고 있고 hepa cell의 zontaion 정보를 완벽하게 알 수 있다. 그렇다면 sample내에 hepa cell 유전자를 이용해 zonation, 즉 위치정보를 역으로 추정할 수 있다. 그렇게해서 central 쪽에서 나왔을 것이라 생각되는 sample을 봤더니 Rspo3, Wnt9b도 높더라! 하는게 아래 그래프이다.
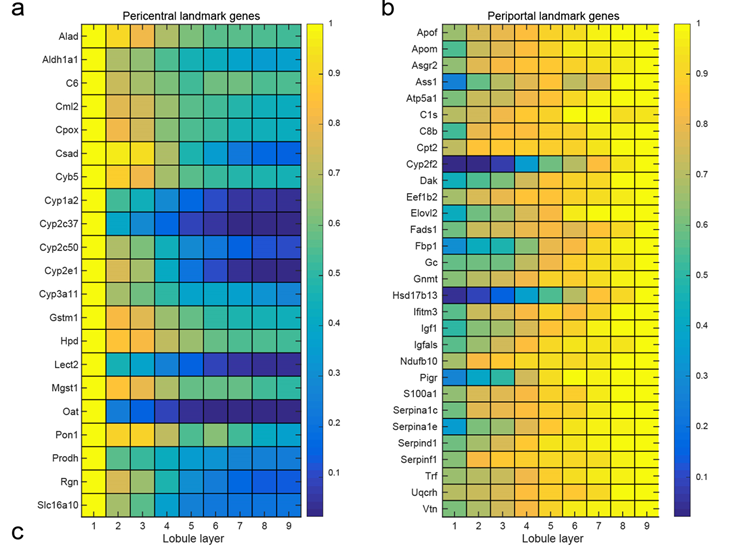
시작할때 hepatoctyte gene atlas는 완벽하게 구축이 되었다고 했는데, 그게 위의 a,b번 panel 이다. 이 유전자를 이용해서 η value 를 구한다. η value가 0에 가까울 수록 central vein, 1에 가까울 수록 portal vein에 가깝다고 생각하면 된다. 예를들어, η value 가 0.5라면 CV 와 PV의 중간 layer 5 혹은 6에 있을 확률이 제일 높다고 보면 된다. η value를 구하는 법은 통계학적인 기법으로 풀어내야하는 내용이라 일단 패스 했다.
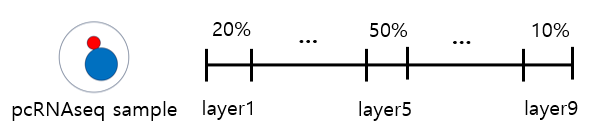
η value 값을 기반으로 각 layer 에 속할 확률을 구한다. 예를어 η value 가 0.5면 layer5에 속할 확률이 가장 높고 양쪽으로 내려갈 갈수록 낮아지는 구조이다. 이 방법역시 통계학이 세게 들어갔기 때문에 여기까지만 이해하고 넘어갔다.
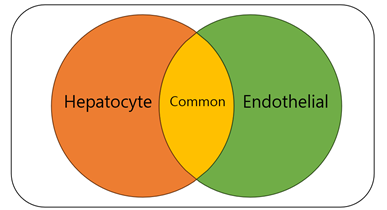

다시 벤다이어그램으로 돌아와서 전체 유전자에서 hepatocyte 유전자를 제거하고, 그 외 QC조건을 pass 한 1,303을 찾아낸다. 이 gene 에 대해서 각 layer 발현양을 구해서 발현양상을 확인한다. 오른쪽 table의 Sample 1을 보자. Sample 에 각 layer에 속할 확률을 위해서 구해놨으니 Gene1의 발현양을 각각의확률에 곱하면 각각의 layer 마다 gene1의 발현 기대값을 구할 수 있다. 이제 layer1에서 Sample1~N 을 모두 합하고 발현확률을 더한 값으로 나눠주면 Gene1의 layer 별 발현 평균치를 구할 수 있다 !! ( 다음 그림의 panel a) 1,303개의 유전자 모두를 테스트해서 gradation을 보이는 475개 유전자를 찾아냈다.
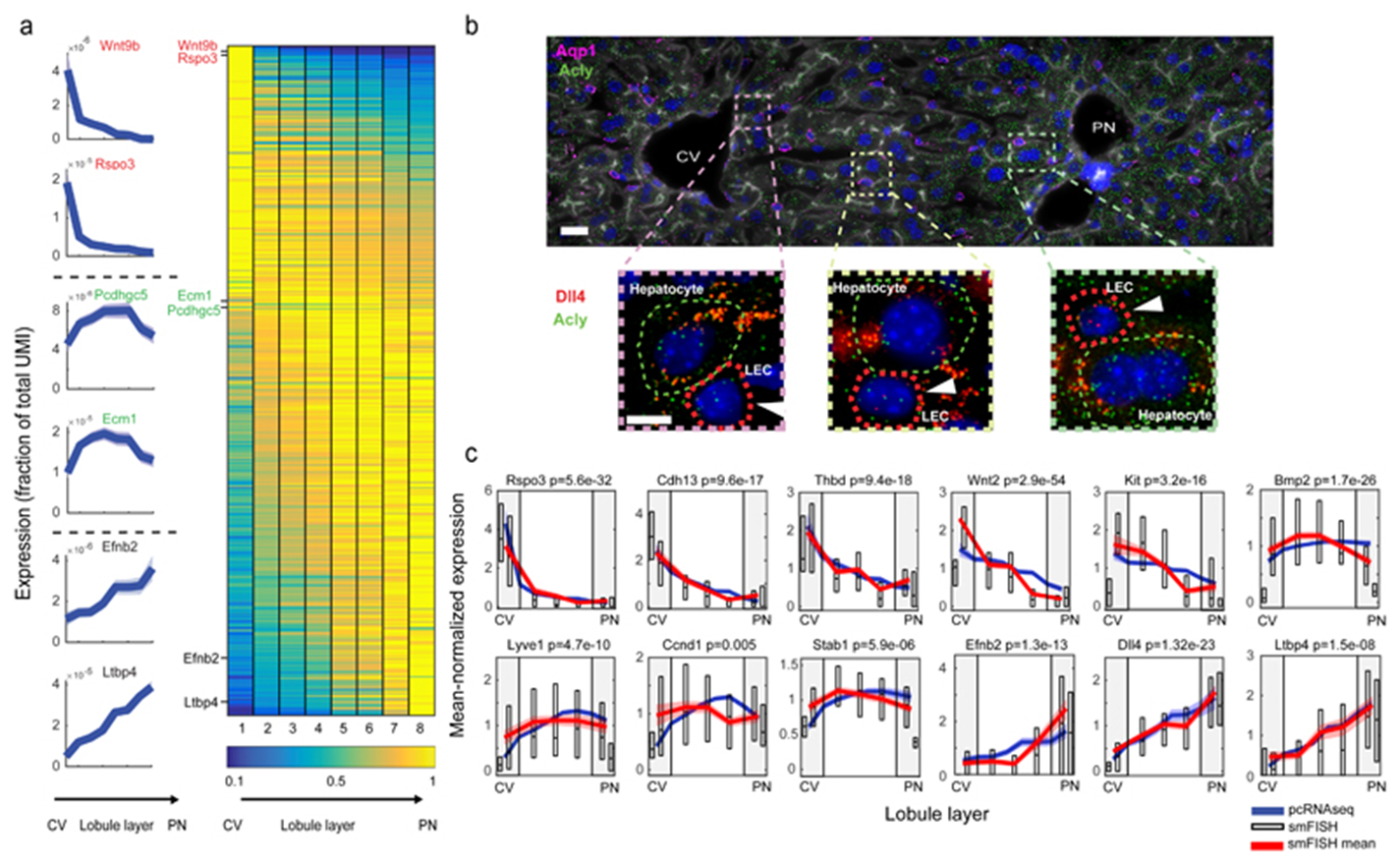
LEC zonated gene 475개가 panel a에 나열되어 있고 왼쪽에 대표6개 유전자 그래프를 보여주고 있다. b에서는 Dll4을 smFISH로 validation 한 결과 pcRNAseq 결과와 일치하는 것을 확인할 수 있다.
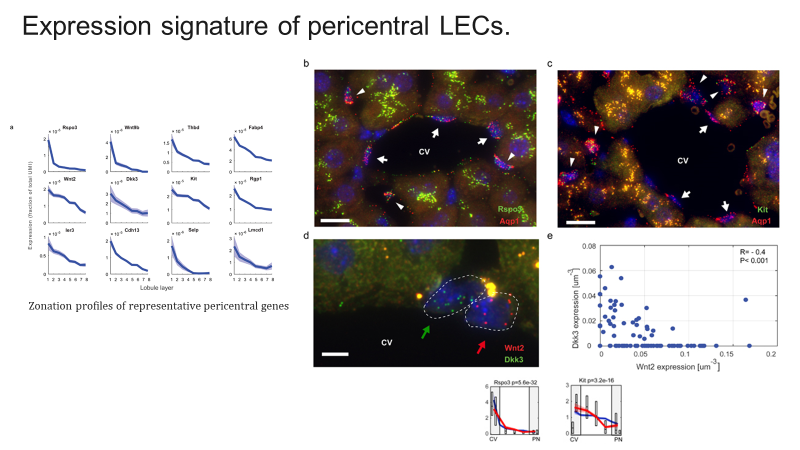
마찬가지로 validation의 연장선인거 같은데 Rapo3 와 Kit유전자를 smFISH 로 확인한 결과이다. Rspo3에서는 1,2 layer 모두에서 높게 발현한 것을 확인했고, 한가지 발견은 Dkk3 와 Wnt2가 반비례관계로 세포에서 발현하는 것을 발견했다. d 패널에서 Wnt2 high, Dkk3 high cell 이 다른것을 확인할 수 있다.
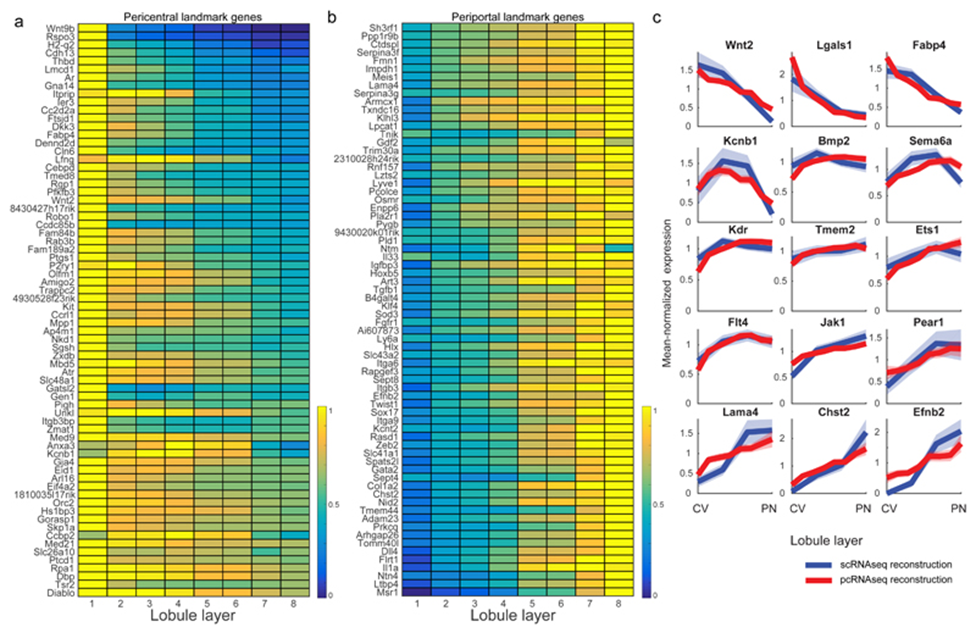
pcRNAseq 에서 찾아낸 LEC specific gene 을 central , portal vein으로 각각 70개씩 추려서 gene panel을 만든 것이다. c에서는 scRNAseq과 pcRNAseq 결과가 같은 경향으로 보인다는 것.
저널 미팅 끝!


